今天通过分析实验结果,完善了之前的预处理代码,借此博文分享给大家,另外
点击此处
可以查看预处理的完整代码,欢迎大家一起来完善。
最重要的是,欢迎大家的批评指正,您的建议和意见将会是我成长源泉。
接下来开始进入正题。
灰度值转换为HU值
基本知识
首先,我们要明确,
CT值
和
灰度值
属于不同的概念。
CT值属于医学领域的概念,通常称亨氏单位(hounsfield unit ,HU),反映了组织对X射线吸收程度。
灰度值属于计算机领域的概念,指的是单个像素点的亮度,灰度值越大表示越亮。范围一般从0到255,白色为255 ,黑色为0,故黑白图片也称灰度图像。
其次,我们了解两种最常用的医学图像数据格式:
DICOM类型
和
NIFTI类型
DICOM类型的文件后缀为
dcm
,常用的python处理库有:SimpleITK, pydicom库 ;
NIFTI类型型的文件后缀为
nii或nii.gz
(压缩格式),常用的python处理库有:SimpleITK, nibabel库。
对于nii格式的图片,SimpleITK,nibabel中常用的api接口,都会自动的进行上述转化过程,即取出来的值已经是Hu了。(除非专门用nib.load(‘xx’).dataobj.get_unscaled()或者itk.ReadImage(‘xx’).GetPixel(x,y,z)才能取得原始数据)
对于dcm格式的图片,SimpleITK, pydicom常用的api接口都不会将原始数据自动转化为Hu!!(itk snap软件读入dcm或nii都不会对数据进行scale操作)
以下我用SimpleITK读取的NIFTI文件,打印出来的最大值和最小值范围,很明显,它们已经是Hu值了,而非灰度值,无需做Hu值变换。
0 all:( -3024.0 , 1410.0 ) bg:( -3024.0 , 1410.0 ) liver:( -604.0 , 333.0 ) tumor:( -0.0 , 160.0 )
公式与代码
HU = pixel_val*slope+ intercept
其中,slope,intercept可以从元数据中读取
def get_pixels_hu(ct_array,slope,intercept):
if slope != 1:
ct_array = slope * ct_array
ct_array += intercept
return ct_array
CT图像窗宽窗位调整
注意:
- 窗宽窗位是CT图像特有的概念,MRI图像中可没有此概念;
- CT 图像必须先转换成 HU值再做窗宽窗位调整。
基本知识
医学图像领域的窗口技术,包括窗宽(window width)和窗位(window center),用于选择感兴趣的CT值范围。因为各种组织结构或病变具有不同的CT值,因此欲显示某一组织结构细节时,应选择适合观察该组织或病变的窗宽和窗位,以获得最佳显示。
窗宽
是CT图像上显示的CT值范围,在此CT值范围内的组织和病变均以不同的模拟灰度显示。而CT值高于此范围的组织和病变,无论高出程度有多少,均以白影显示,不再有灰度差异; 反之,低于此范围的组织结构,不论低的程度有多少,均以黑影显示,也无灰度差别。增大窗宽,则图像所示CT值范围加大,显示具有不同密度的组织结构增多,但各结构之间的灰度差别减少。减小窗宽,则显示的组织结构减少,然而各结构之间的灰度差别增加。如观察脑质的窗宽常为-15~+85H,即密度在-15 ~+85H范围内的各种结构如脑质和脑脊液间隙均以不同灰度显示。而高于+85H的组织结构如骨质几颅内钙化,其间虽有密度差,但均以白影显示,无灰度差别;而低于-15H组织结构如皮下脂肪及乳突内气体均以黑影显示,其间也无灰度差别。
窗位
是窗的中心位置,同样的窗宽,由于窗位不同,其所包括CT值范围的CT
值也有差异。例如窗宽同为100H,当窗位为0H时,其CT值范围为-50 ~ +50H ; 如窗位为+35H时,则CT值范围为-15~+85H。通常,欲观察某以组织结构及发生的病变,应以该组织的CT值为窗位。例如脑质CT值约为+35H,则观察脑组织及其病变时,选择窗位以+35H为妥。
设定正确的窗宽窗位
应用场景:用神经网络分割liver 和liver tumor,对训练数据进行预处理。
基本思路:
- 用liver和tumor的mask抠出liver和tumor部分;
-
统计相应的最大值和最小值,并用其计算窗宽窗位。
优点:每个case的窗宽窗位依据case量身定做。相较于整个数据集用统一的窗宽窗位,或者采用cut off,有无可比拟的优点。
代码
import SimpleITK as sitk
import numpy as np
import os
def saved_preprocessed(savedImg,origin,direction,xyz_thickness,saved_name):
newImg = sitk.GetImageFromArray(savedImg)
newImg.SetOrigin(origin)
newImg.SetDirection(direction)
newImg.SetSpacing((xyz_thickness[0], xyz_thickness[1], xyz_thickness[2]))
sitk.WriteImage(newImg, saved_name)
def window_transform(ct_array, windowWidth, windowCenter, normal=False):
"""
return: trucated image according to window center and window width
and normalized to [0,1]
"""
minWindow = float(windowCenter) - 0.5*float(windowWidth)
newimg = (ct_array - minWindow) / float(windowWidth)
newimg[newimg < 0] = 0
newimg[newimg > 1] = 1
if not normal:
newimg = (newimg * 255).astype('uint8')
return newimg
ct_path = 'G:\LITS\Training_dataset'
saved_path = 'C:\\Users\\Cerry\\Desktop\\wl'
name_list = ['volume-0.nii']
for name in name_list:
ct = sitk.ReadImage(os.path.join(ct_path,name))
origin = ct.GetOrigin()
direction = ct.GetDirection()
xyz_thickness = ct.GetSpacing()
ct_array = sitk.GetArrayFromImage(ct)
seg_array = sitk.GetArrayFromImage(sitk.ReadImage(os.path.join(ct_path,name.replace('volume','segmentation'))))
seg_bg = seg_array == 0
seg_liver = seg_array >= 1
seg_tumor = seg_array == 2
ct_bg = ct_array * seg_bg
ct_liver = ct_array * seg_liver
ct_tumor = ct_array * seg_tumor
liver_min = ct_liver.min()
liver_max = ct_liver.max()
tumor_min = ct_tumor.min()
tumor_max = ct_tumor.max()
#by liver
liver_wide = liver_max - liver_min
liver_center = (liver_max + liver_min)/2
liver_wl = window_transform(ct_array, liver_wide, liver_center, normal=True)
saved_name = os.path.join(saved_path,'liver_wl_1.nii')
saved_preprocessed(liver_wl, origin, direction, xyz_thickness, saved_name)
#by tumor (recommended)
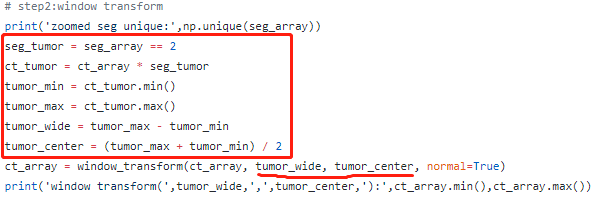
tumor_wide = tumor_max - tumor_min
tumor_center = (tumor_max + tumor_min) / 2
tumor_wl = window_transform(ct_array, tumor_wide, tumor_center, normal=True)
saved_name = os.path.join(saved_path, 'tumor_wl_1.nii')
saved_preprocessed(tumor_wl, origin, direction, xyz_thickness, saved_name)
实验过程
原始CT图像

用liver的最大值和最小值计算窗宽窗位

用tumor的最大值和最小值计算窗宽窗位

由实验结果可知,用tumor的最大值和最小值计算窗宽窗位,所得结果最好。
Last but not least!!!
一种错误的实现方式,具体糟糕的实验结果如下所示:
// 先算图像的最大和最小值
min = (2*window_center - window_width)/2.0 + 0.5;
max = (2*window_center + window_width)/2.0 + 0.5;
for (i = 0; i < nNumPixels; i++)
disp_pixel_val = (pixel_val - min)*255.0/(double)(max - min);

参考
如何设定正确的窗宽窗位,附正常人体组织CT值
医学影像“调窗”(window-leveling)的算法
CT值与灰度值的总结
常见医疗扫描图像处理步骤