2021.05.05更新:
将基因名称转化为基因ID
上一次在这个平台上写文章居然已经是5年前了,毕竟研究生阶段没有主攻数据处理,让自己少了很多IT属性。废话少说,今天记录一下将基因ID转化为基因名称的操作办法。
在拿到公司的基因测序数据后,不同的测序公司给出的数据也是不同的,有时候会遇到一个问题就是测序公司给出的分析报告中,GO富集以及KEGG通路通常直接以基因ID的形式给客户,而导师要求送审文章的附件要显示为基因名称。

尽管在拿到的测序数据中有所有原始数据的基因ID以及对应的基因名称的list,但一一对应转换显然不太现实。因此,读研期间,我用Excel制作了一个转换工作台来完成这项工作,没想到毕业之后,师弟师妹仍然有这个需求。于是我打算写这个文章,将这个简单的处理文档分享给大家。
转换总共包括基因格式处理、匹配基因ID与基因name、生成基因name、清洗空白域、添加分隔符、粘贴文本格式、合并基因以及粘贴文本格式并清洗分隔符8个步骤,在excel文档中每个sheet都为1个步骤。看起来似乎有些麻烦,但每一个步骤涉及的操作只有1步,可以说讲转化步骤完全拆解了。
step1.分列处理
第一步首先将待转化的基因ID列表复制粘贴到step1表格中,GO分析和KEGG基因ID列表一般为单列,而后面步骤转化每个基因ID时都需要每个基因ID单独占一个单元格。因此,第一步我们需要将单列的基因ID进行分列处理。
选中该列数据,在上方菜单栏中找到【数据】,【分列】→【分隔符号】→【分号】(根据实际情况选择)→【完成】,即可将每一个基因ID放入对应的单元格中。

step2.匹配基因ID与基因name
第二步很简单,既然要转换,就需要一个“字典”,给后面的转化做参考,我们在测序文档中将原始数据的基因ID以及对应的基因name两列复制粘贴到step2的表格里就可以了。需要注意的是“字典”的基因ID一定要全,确保你转换的基因ID都被包含在了“字典”里面。

step3.生成基因name
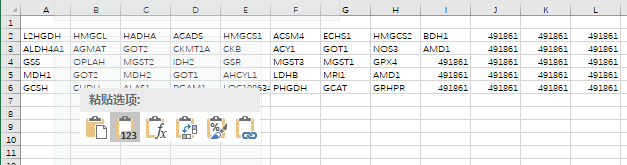
如果你的前两步都没有问题,基因ID已经分列成功,“字典”内容完整,那么到step3的时候,你会惊奇地发现你的基因name已经生成出来了!转换的函数其实不难,一个IFERROR函数内嵌一个VLOOKUP函数,IFERROR函数判定是否有基因ID需要匹配或者是基因ID是否包含于“字典”中。value_if_error值(491861)可以自己设定,只要保证value_if_error值不会出现在基因name中就行,避免在清洗环节把基因name修改了,后面的步骤则是将基因name还原成step1中的单列格式。

step4.清洗空白域
这一步稍微有些复杂,需要我们将steo3生成的基因name所在行复制到step4当中,粘贴时,选择
粘贴 值(按钮角标是123),不要直接Ctrl+V,这样在step4当中的基因name才不会显示输出错误。然后选中所有基因name行,在菜单栏【开始】→【查找和选择】→【替换】,将
value_if_error值(491861)删除掉(替换为一栏留白即可),即可清洗空白域
step5.添加分隔符
第五步是在每个基因name后面通过“&”符号添加分隔符,这样方便合并,这一步只需要将带分隔符的基因name所在行复制到下一步就可以了。
step6.粘贴为文本格式
第六步与step4相同,粘贴时选择粘贴 值,作为下一步的合并数据。
step7.合并基因name
使用PHONETIC函数将同一行单元格内的所有基因name合并到第一列的单元格中,如果你的第六步没有问题,到这里可以直接得到合并集,我们马上就要成功了!

step8.清洗分隔符
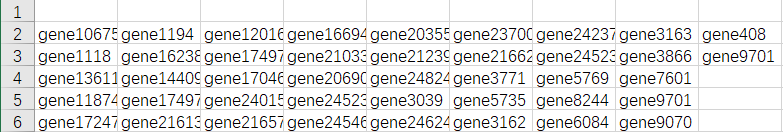
最后一步,将step7的第一列以
值
的形式
粘贴到
最后一步,在菜单栏选择
【开始】→【查找和选择】→【替换】,将 ,,(双逗号)
替换为空白,这样,最后一个基因后面的分隔符会被清除掉,而基因name之间的分隔符仍然会被保留。
来看下替换前后的样子:

以上就是基因ID替换为基因name的操作流程,大家可以根据自己的实际情况对文档进行修改,工作文档直接在下方资源信息处下载即可。
欢迎生信爱好者加群交流讨论,遇见二维码过期可添加VX:bbplayer2021 ,备注 申请加入生信交流群。
