R语言也有“一步到位”的函数,如prcomp()和princomp(),基本上都是输入数据直接出结果。为了理解PCA的原理,我们利用自编函数的方法进行学习。

主成分分析详解
主成分分析过程分解
1.数据标准化
为了统一数据的量纲并对数据进行中心化,在主成分分析之前往往需要对原始数据进行标准化。下面以R语言自带的iris范例数据集为例,探索一下主成分分析的具体过程。
#将R自带的范例数据集iris储存为变量data;
data<-iris
head(data)
#对原数据进行z-score归一化;
dt<-as.matrix(scale(data[,1:4]))
head(dt)

2.计算相关系数(协方差)矩阵
既然主成分分析主要是选取解释变量方差最大的主成分,故先需要计算变量两两之间协方差,根据协方差与方差的关系,位于协方差矩阵对角线上的数值即为相应变量的方差。此外,由于对数据进行了Z-score归一化(变量的均值为0,标准差为1);因此,根据相关系数的计算公式可知,此时相关系数其实等于协方差。
#计算相关系数矩阵;
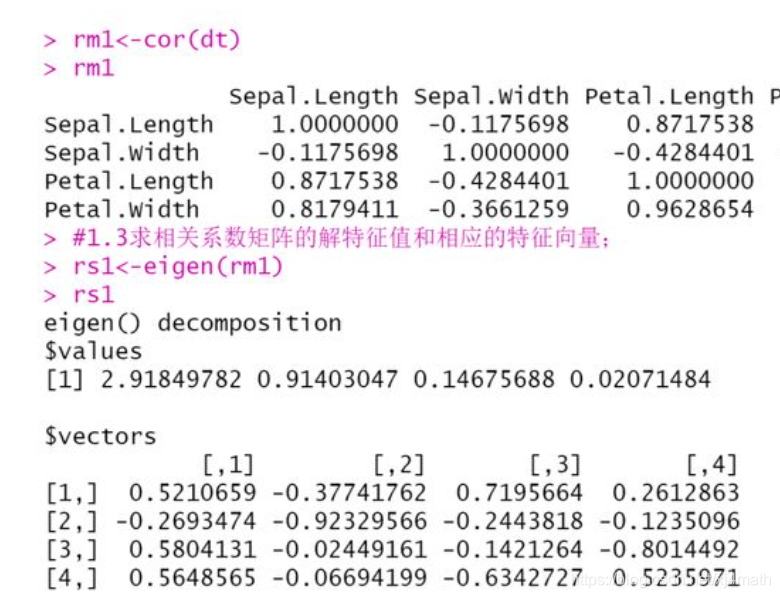
rm1<-cor(dt)
rm1
3.求解特征值和相应的特征向量
rs1<-eigen(rm1)
rs1
#提取结果中的特征值,即各主成分的方差;
val <- rs1$values
#换算成标准差(Standard deviation);
(Standard_deviation <- sqrt(val))
#计算方差贡献率和累积贡献率;
(Proportion_of_Variance <- val/sum(val))
(Cumulative_Proportion <- cumsum(Proportion_of_Variance))

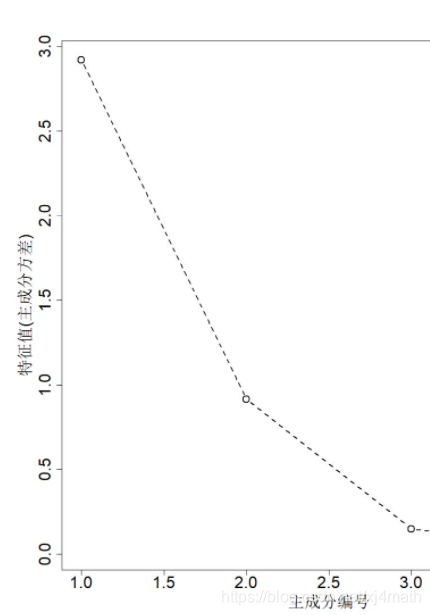
#碎石图绘制;
par(mar=c(6,6,2,2))
plot(rs1$values,type="b",
cex=2,
cex.lab=2,
cex.axis=2,
lty=2,
lwd=2,
xlab = "主成分编号",
ylab="特征值(主成分方差)")
4.计算主成分得分
#提取结果中的特征向量(也称为Loadings,载荷矩阵);
(U<-as.matrix(rs1$vectors))
#进行矩阵乘法,获得PC score;
PC <-dt %*% U
colnames(PC) <- c("PC1","PC2","PC3","PC4")
head(PC)

5.绘制主成分散点图
#将iris数据集的第5列数据合并进来;
df<-data.frame(PC,iris$Species)
head(df)
#载入ggplot2包;
library(ggplot2)
#提取主成分的方差贡献率,生成坐标轴标题;
xlab<-paste0("PC1(",round(Proportion_of_Variance[1]*100,2),"%)")
ylab<-paste0("PC2(",round(Proportion_of_Variance[2]*100,2),"%)")
#绘制散点图并添加置信椭圆;
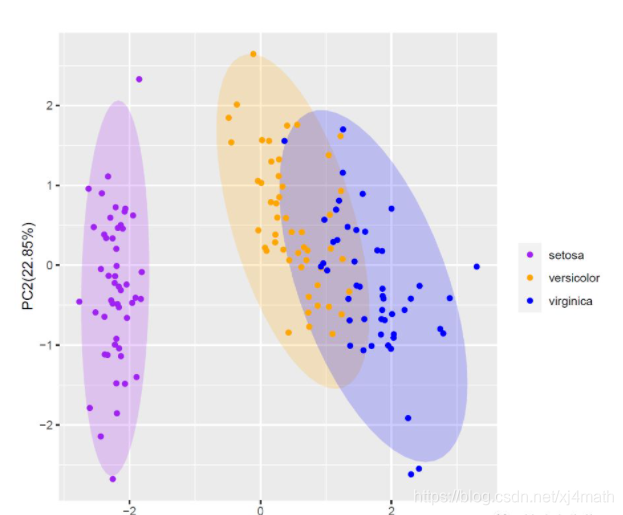
p1<-ggplot(data = df,aes(x=PC1,y=PC2,color=iris.Species))+
stat_ellipse(aes(fill=iris.Species),
type ="norm", geom ="polygon",alpha=0.2,color=NA)+
geom_point()+labs(x=xlab,y=ylab,color="")+
guides(fill=F)
p1
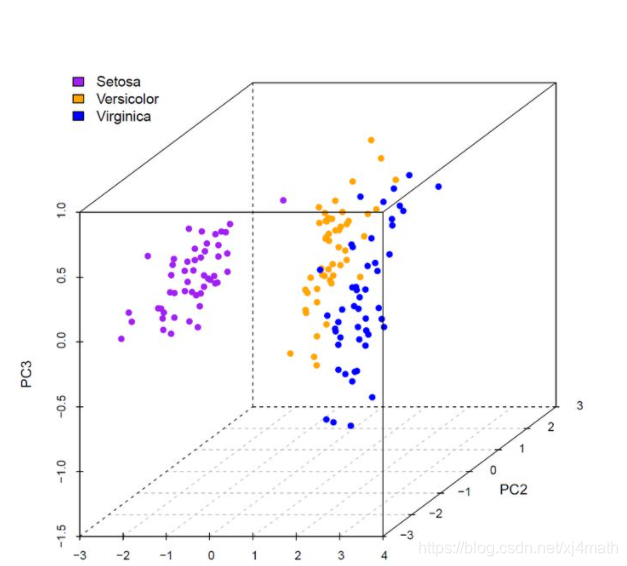
下面,尝试使用3个主成分绘制3D散点图。
#载入scatterplot3d包;
library(scatterplot3d)
color = c(rep('purple',50),rep('orange',50),rep('blue',50))
scatterplot3d(df[,1:3],color=color,
pch = 16,angle=30,
box=T,type="p",
lty.hide=2,lty.grid = 2)
legend("topleft",c('Setosa','Versicolor','Virginica'),
fill=c('purple','orange','blue'),box.col=NA)
6 自编函数实现PCA总代码
#将R自带的范例数据集iris储存为变量data;
data<-iris
head(data)
#对原数据进行z-score归一化;
dt<-as.matrix(scale(data[,1:4]))
head(dt)
#计算相关系数矩阵;
rm1<-cor(dt)
rm1
#求特征值和特征向量
rs1<-eigen(rm1)
rs1
#提取结果中的特征值,即各主成分的方差;
val <- rs1$values
#换算成标准差(Standard deviation);
(Standard_deviation <- sqrt(val))
#计算方差贡献率和累积贡献率;
(Proportion_of_Variance <- val/sum(val))
(Cumulative_Proportion <- cumsum(Proportion_of_Variance))
#碎石图绘制;
par(mar=c(6,6,2,2))
plot(rs1$values,type="b",
cex=2,
cex.lab=2,
cex.axis=2,
lty=2,
lwd=2,
xlab = "主成分编号",
ylab="特征值(主成分方差)")
#计算主成分
#提取结果中的特征向量(也称为Loadings,载荷矩阵);
(U<-as.matrix(rs1$vectors))
#进行矩阵乘法,获得PC score;
PC <-dt %*% U
colnames(PC) <- c("PC1","PC2","PC3","PC4")
head(PC)
#绘制主成分散点图
#将iris数据集的第5列数据合并进来;
df<-data.frame(PC,iris$Species)
head(df)
#载入ggplot2包;
library(ggplot2)
#提取主成分的方差贡献率,生成坐标轴标题;
xlab<-paste0("PC1(",round(Proportion_of_Variance[1]*100,2),"%)")
ylab<-paste0("PC2(",round(Proportion_of_Variance[2]*100,2),"%)")
#绘制散点图并添加置信椭圆;
p1<-ggplot(data = df,aes(x=PC1,y=PC2,color=iris.Species))+
stat_ellipse(aes(fill=iris.Species),
type ="norm", geom ="polygon",alpha=0.2,color=NA)+
geom_point()+labs(x=xlab,y=ylab,color="")+
guides(fill=F)
p1
#载入scatterplot3d包;
library(scatterplot3d)
color = c(rep('purple',50),rep('orange',50),rep('blue',50))
scatterplot3d(df[,1:3],color=color,
pch = 16,angle=30,
box=T,type="p",
lty.hide=2,lty.grid = 2)
legend("topleft",c('Setosa','Versicolor','Virginica'),
fill=c('purple','orange','blue'),box.col=NA)
使用现成函数完成主成分分析
R中最常见的两个PCA函数:prcomp()和princomp()。了解了主成分分析的具体步骤后,接下来使用这两个“一步到位”的函数进行验证以上分析过程是否正确。
1.prcomp()函数
#scale. = TRUE表示分析前对数据进行归一化;
com1 <- prcomp(data[,1:4], center = TRUE,scale. = TRUE)
summary(com1)
2.princomp()函数
如果使用princomp()函数,需要先做归一化,princomp()函数并无数据标准化相关的参数。且默认使用covariance matrix,得到的结果与使用相关性矩阵有细微差异(如下);原因是根据相关系数公式可知,归一化后的相关性系数近乎等于协方差。
com2 <- princomp(dt,cor = T)
summary(com2)
com3 <- princomp(dt)
summary(com3)
注意,princomp()函数只适用于行数大于列数的矩阵,否则会报错:Error in princomp.default(dt) : ‘princomp’只能在单位比变量多的情况下使用。
通过比较分析得到Standard deviation、Proportion of Variance、Cumulative Proportion发现,3种主成分分析的方法得到的结果完全一致。
3.PCA结果可视化
以prcomp()函数的结果为例,提取不同记录的PC1~PC4 score数值,即点的横纵坐标值。
#提取PC score;
df1<-com1$x
head(df1)
#将iris数据集的第5列数据合并进来;
df1<-data.frame(df1,iris$Species)
head(df1)
同上,接下来对分析结果进行可视化,即绘制散点图并添加置信椭圆。
#提取主成分的方差贡献率,生成坐标轴标题;
summ<-summary(com1)
xlab<-paste0("PC1(",round(summ$importance[2,1]*100,2),"%)")
ylab<-paste0("PC2(",round(summ$importance[2,2]*100,2),"%)")
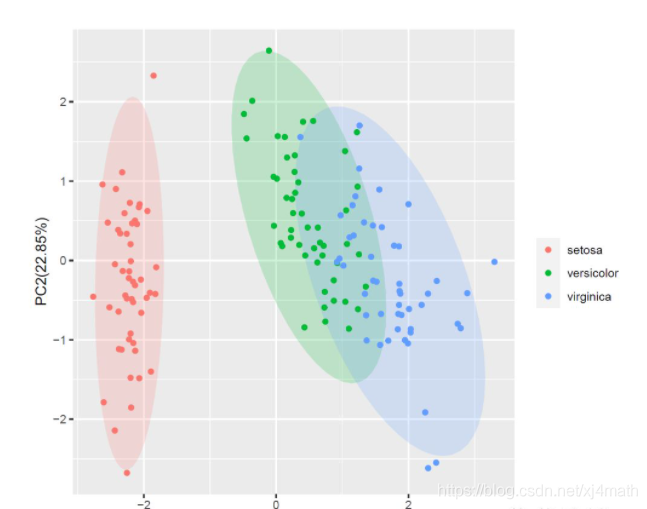
p2<-ggplot(data = df1,aes(x=PC1,y=PC2,color=iris.Species))+
stat_ellipse(aes(fill=iris.Species),
type = "norm", geom ="polygon",alpha=0.2,color=NA)+
geom_point()+labs(x=xlab,y=ylab,color="")+
guides(fill=F)
p2+scale_fill_manual(values = c("purple","orange","blue"))+
scale_colour_manual(values = c("purple","orange","blue"))
绘制效果如下: