1、二叉树的右视图
给定一个二叉树的根节点
root
,想象自己站在它的右侧,按照从顶部到底部的顺序,返回从右侧所能看到的节点值。
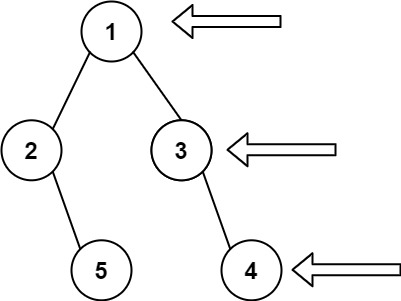
示例1:
输入: [1,2,3,null,5,null,4]
输出: [1,3,4]
示例 2:
输入: [1,null,3]
输出: [1,3]
示例 3:
输入: []
输出: []
#include <bits/stdc++.h>
using namespace std;
struct TreeNode
{
int val;
TreeNode *left;
TreeNode *right;
TreeNode(int x) : val(x), left(NULL), right(NULL) {}
};
class Solution
{
public:
vector<int> rightSideView(TreeNode *root)
{
vector<int> ret;
queue<TreeNode *> queues[2];
if (root != NULL)
queues[0].push(root);
int i = 0, j = 1, tmp;
TreeNode *p;
while (!queues[0].empty() || !queues[1].empty())
{
while (!queues[i].empty())
{
p = queues[i].front();
queues[i].pop();
if (p->left)
queues[j].push(p->left);
if (p->right)
queues[j].push(p->right);
tmp = p->val;
}
ret.push_back(tmp);
i = (i + 1) % 2;
j = (j + 1) % 2;
}
return ret;
}
};
2、输入一个数查找比它小的元素
一个整型数组有 10 个元素,请先给这 10 个元素赋值,然后随便输入一个数,最后输出数组中比这个数小的所有元素,每个数用空格隔开,如果没有找到,则输出 not find。
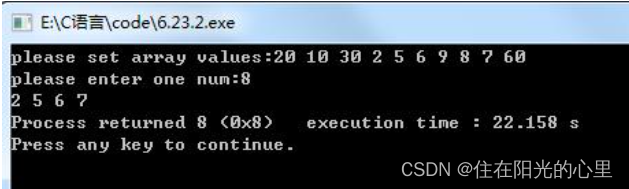
#include <stdio.h>
int main()
{
int a[10], i, n;
int isfind = 0;
printf("please set array values:");
for (i = 0; i < 10; i++)
scanf("%d", &a[i]);
printf("please enter one num:");
scanf("%d", &n);
for (i = 0; i < 10; i++)
{
if(a[i] < n){
isfind = 1;
printf("%d", a[i]);
}
}
if (isfind)
printf("\n");
else
printf("not find\n");
return 0;
}
3、重复的DNA序列
所有 DNA 都由一系列缩写为
'A'
,
'C'
,
'G'
和
'T'
的核苷酸组成,例如:
"ACGAATTCCG"
。在研究 DNA 时,识别 DNA 中的重复序列有时会对研究非常有帮助。编写一个函数来找出所有目标子串,目标子串的长度为 10,且在 DNA 字符串
s
中出现次数超过一次。示例 1:
输入:s = “AAAAACCCCCAAAAACCCCCCAAAAAGGGTTT”
输出:[“AAAAACCCCC”,”CCCCCAAAAA”]
示例 2:
输入:s = “AAAAAAAAAAAAA”
输出:[“AAAAAAAAAA”]
提示:
0 <= s.length <= 105
s[i]
为
'A'
、
'C'
、
'G'
或
'T'
#include <bits/stdc++.h>
using namespace std;
class Solution
{
public:
vector<string> findRepeatedDnaSequences(string s)
{
std::map<std::string, int> word_map;
std::vector<std::string> result;
for (int i = 0; i < s.length(); i++)
{
std::string word = s.substr(i, 10);
if (word_map.find(word) != word_map.end())
{
word_map[word] += 1;
}
else
{
word_map[word] = 1;
}
}
std::map<std::string, int>::iterator it;
for (it = word_map.begin(); it != word_map.end(); it++)
{
if (it->second > 1)
{
result.push_back(it->first);
}
}
return result;
}
};