用PCA降维,后输出降维后每一列的贡献率,各列贡献相加为1.
// An highlighted block
if __name__ == "__main__":
iris = datasets.load_iris() # 获取鸢尾花数据集Dick
X=iris["data"]#训练数据
Y=iris["target"]#类别
pca = PCA(n_components=2) # 降到2维
pca.fit(X) # 训练
x = pca.fit_transform(X) # 降维后的数据
print(pca.explained_variance_ratio_) # 输出贡献率
基于鸢尾花数据画图,用PCA降维到两维后
plt.figure(figsize=(10, 5))
# x1_min, x1_max = x[:, 0].min(), x[:, 0].max() # 第0列的范围
# x2_min, x2_max = x[:, 1].min(), x[:, 1].max() # 第1列的范围
# plt.xlim(x1_min, x1_max)
# plt.ylim(x2_min, x2_max)
for i in range(len(Y)):
if Y[i]==0:
plt.plot(x[i, 0], x[i, 1], 'ro')
elif Y[i]==1:
plt.plot(x[i, 0], x[i, 1], 'bo')
else:
plt.plot(x[i, 0], x[i, 1], 'yo')
#gca="get current axis" 设置坐标轴原点
ax = plt.gca()
ax.xaxis.set_ticks_position("bottom")#把四条外边框的下边当做x轴
ax.yaxis.set_ticks_position("left")#把四条外边框的左边当做y轴
ax.spines["bottom"].set_position(("data", 0))#把x轴的位置放到y的0位置
ax.spines["left"].set_position(("data", 0))#把y轴的位置放到x的0位置
plt.grid(True)
plt.xlabel('1st')
plt.ylabel('2nd')
plt.title('Scatter Plot')
plt.show()
效果图如下:

对上图进行
归一化
处理,通过遍历每列feature vector里的每一个数据,将Max和Min的记录下来,并通过Max-Min作为基数(即Min=0,Max=1)进行数据的归一化处理,将每个特征值规约到(0,1)之间。
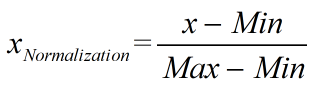
#导入包:from sklearn.preprocessing import MinMaxScaler
#归一化(normalization)
minMax = MinMaxScaler()
x_std = minMax.fit_transform(x)
效果图如下:

标准化
处理:对原始数据的均值(mean)和标准差(standard deviation)进行数据的标准化。经过处理的数据符合标准正态分布,即均值为0,标准差为1,这个方法被广泛的使用在许多机器学习算法中(例如:支持向量机、逻辑回归和类神经网络)。公式如下:
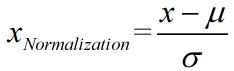
#导入包:from sklearn.preprocessing import StandardScaler
#标准化(Standardization)
scaler = StandardScaler().fit(x)
x_std = scaler .transform(x)#标准化后的数据
print(scaler.mean_)#输出均值
print(scaler.scale_)#输出标准差
版权声明:本文为super_girl_WMM原创文章,遵循 CC 4.0 BY-SA 版权协议,转载请附上原文出处链接和本声明。