软件下载网址:
GSEA (gsea-msigdb.org)
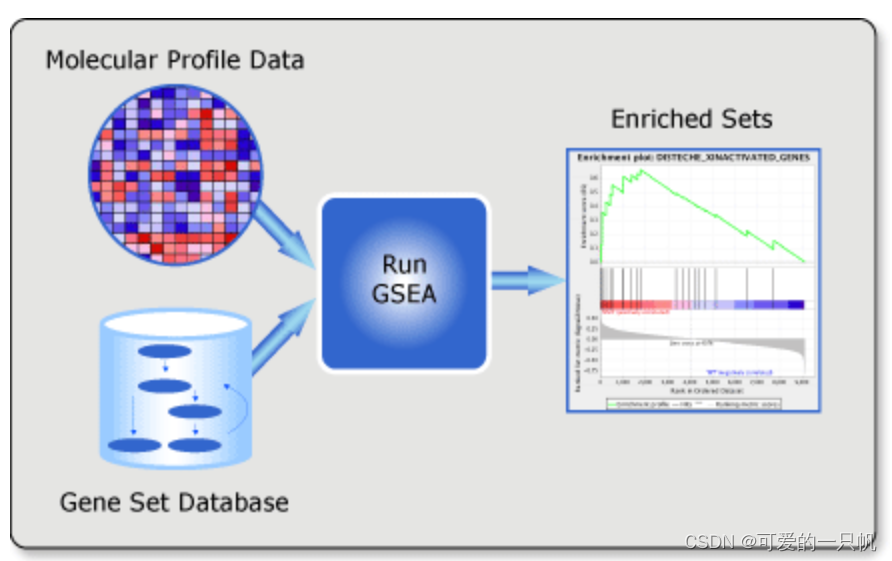
GSEA不需要设置阈值过滤基因,有助于我们从整体通路分析差异。
一.数据准备

1.数据集(tpm_bulk.gct):你需要分析的表达矩阵,建议bulk数据用tpm标准化后的
第一行:#1.2默认的,不用改
第二行:矩阵总共的基因数量和样本数量
第三行及下:你的表达矩阵,Description不能为空,可以是na
可以在excel里处理文件,保存为制表符分隔文件(.txt),直接重命名改后缀为gct即可。

2.样本信息表(DATA_info.cls)

第一行:样品总数、分组数、不用改的数字1
第二行:样品分组名称
第三行:样品分组信息,与.gct文件对应
3.参考基因集(geneset.gmt)
第一列:通路名称
第二列:通路编号
第三列及之后:该通路包含的所有基因,基因名称需要与.gct文件中的基因名称一致
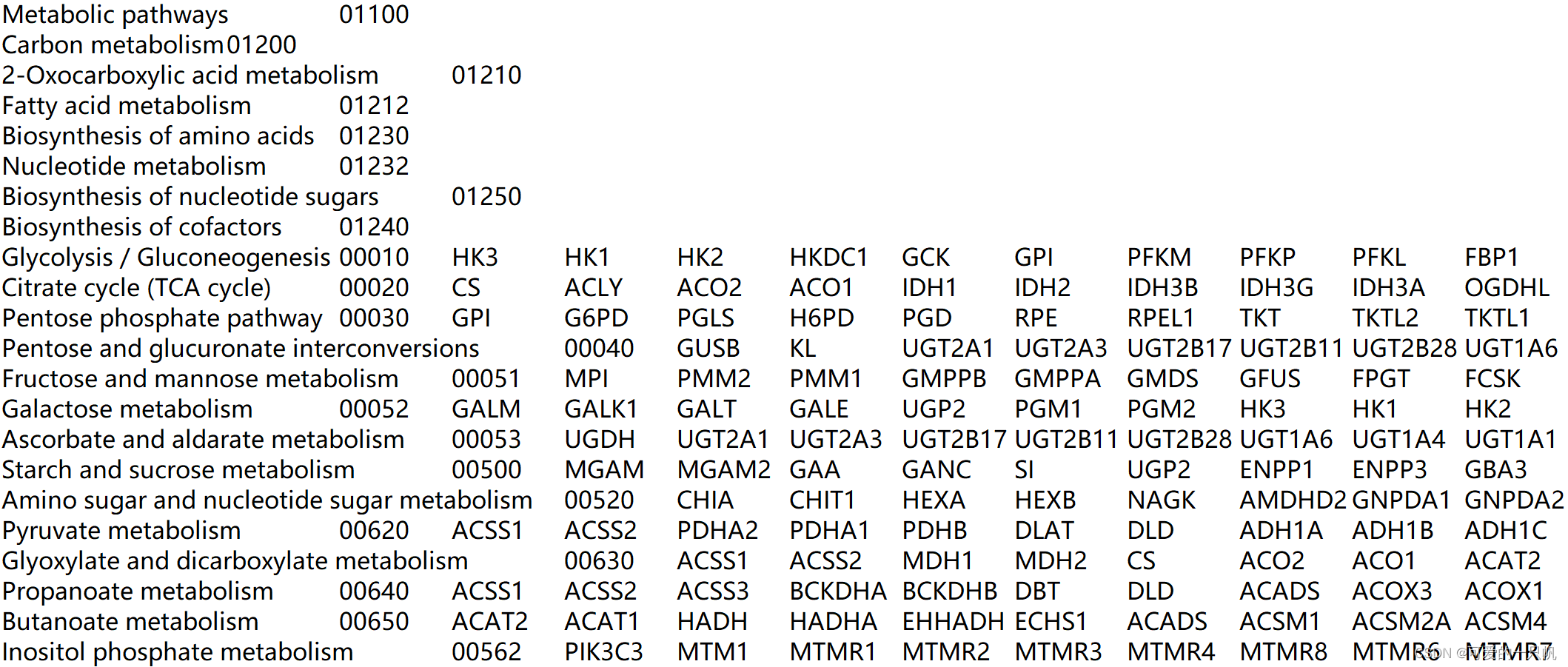
常用的有KEGG和GO基因集,获取基因集的方法:
1)MSigDB官网以及GSEA软件自带的基因集,大部分是有的,但是比KEGG官网上少了很多,官网也有一直在更新,MSigDB官网和GSEA软件没有跟上
2)自己去官网下载制作,生信技能树之前有提到过
3)最近看到python爬取KEGG数据库的,使用更方便,研究中……
二.软件使用
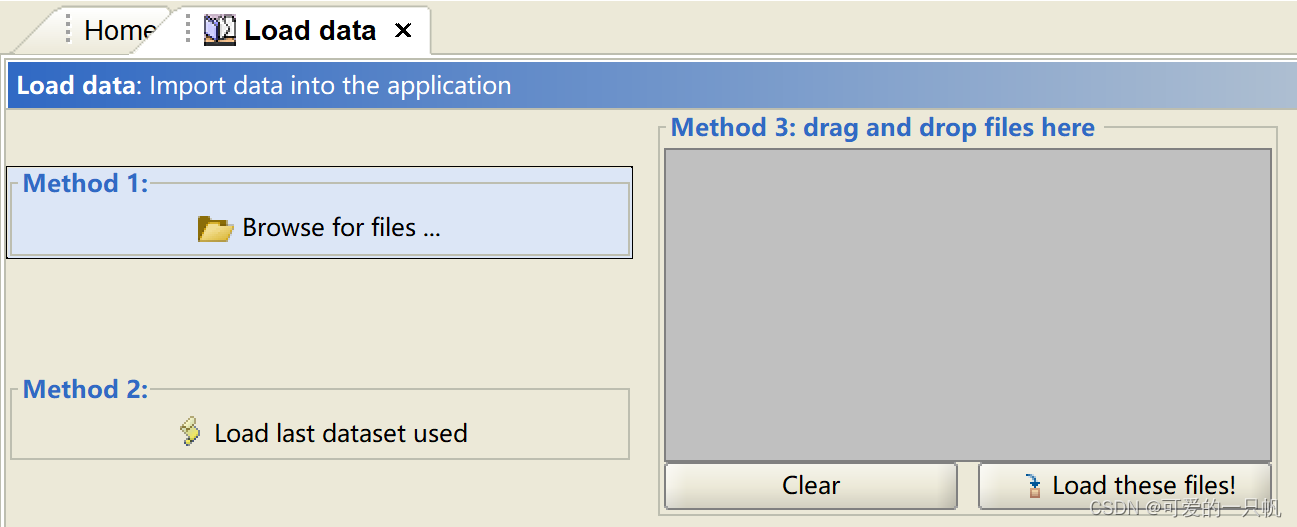
1.上传数据(load data)
直接上传前面准备的三个文件即可,成功了会提示No Error。
2.参数设置
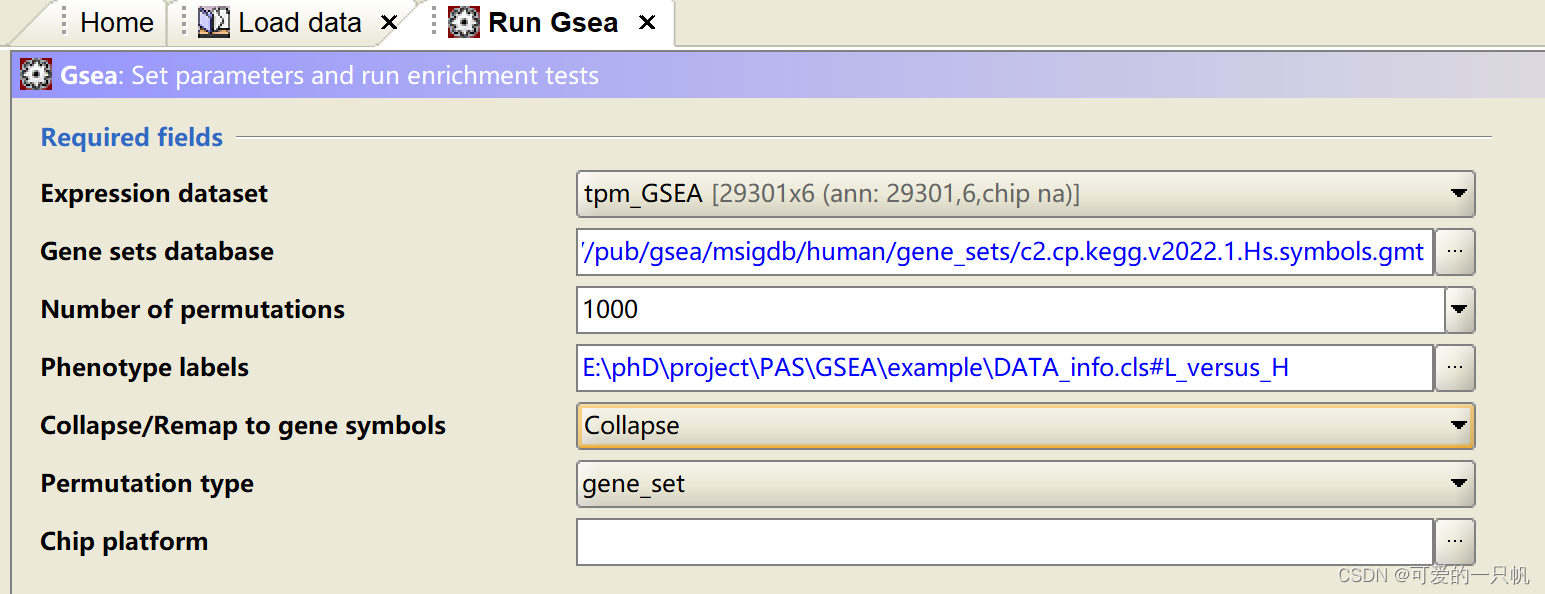
Expression dataset:选择上传的表达矩阵.gct
Gene sets database:选择上传或自带的基因集.gmt
Number of permutations:置换检验的次数,一般为1000
Phenotype labels:需要比较的两组,.cls文件的分组,实验组比对照组
Collapse/Remap to gene symbols:基因symbol转换,文件的基因都是symbol就选No
Permutation type:每组样本数量大于7选phenotype,否则选gene_set
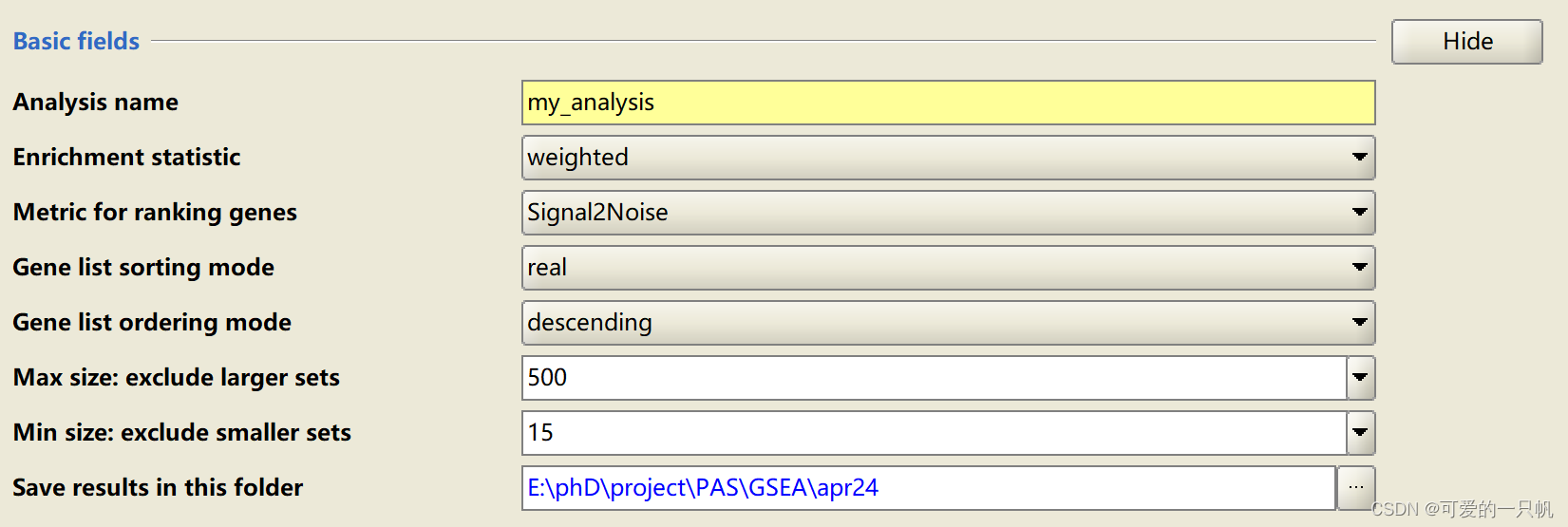
Analysis name:输出的文件名
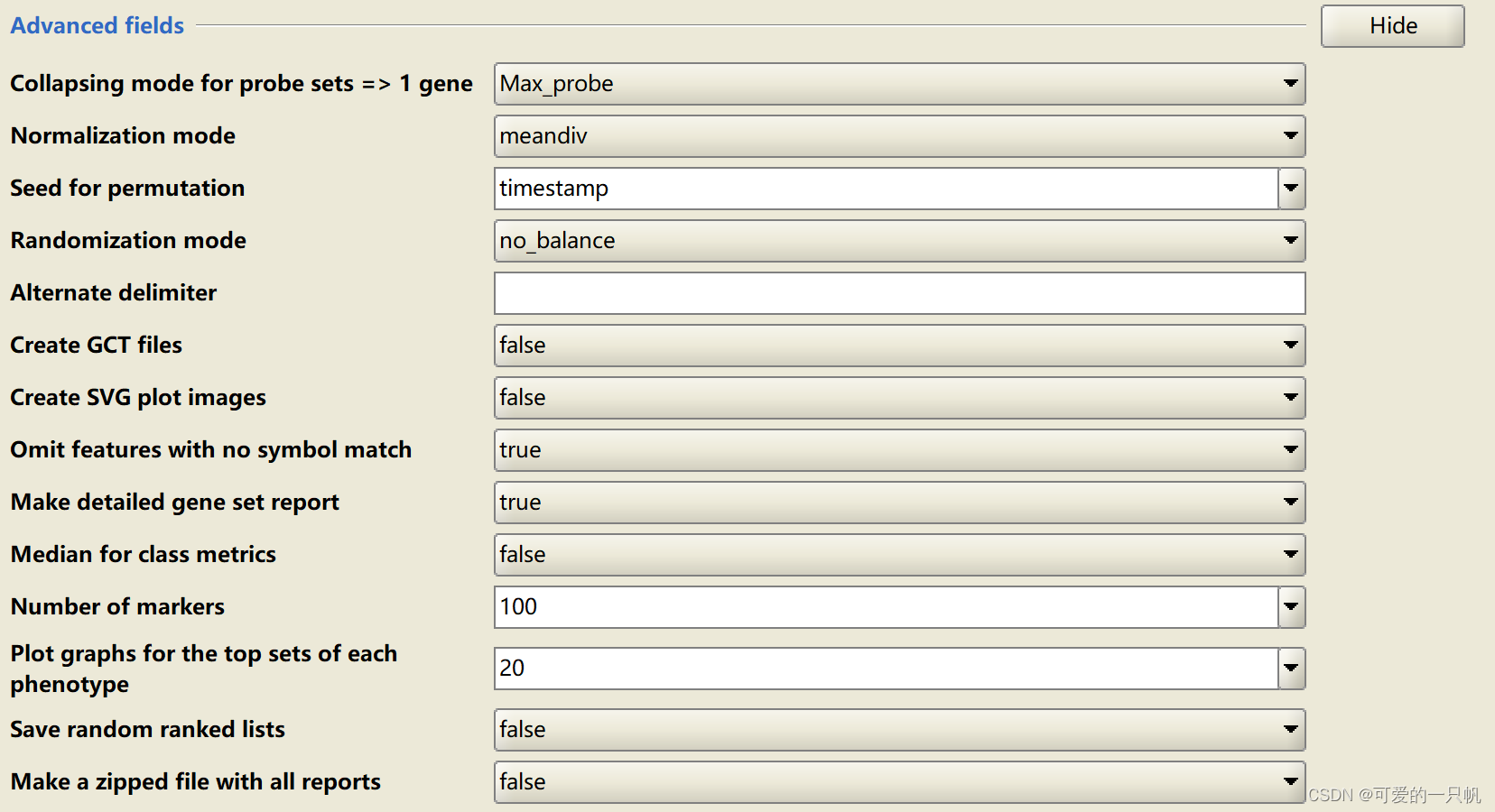
Plot graphs for the top sets of each phenotype:最终画图的数量
3.运行:直接Run

4.查看结果
运行完左侧这里会出现文件名和succes,双击可以打开网页查看结果,一般通过|NES|>1&p-value<5%&FDR q-val<25%筛选结果,如果数量太多可以更严格一些筛选,或者考虑是不是输入的文件有问题。