TCGA
Cox比例风险回归模型临床应用非常广泛,Cox分析得到的结果是可以直接运用到临床应用的,所以这个分析对癌症临床诊断有非常关键的作用,检测高低风险的关键基因,就可以预测病人5年生存率。
Cox比例风险回归模型,简称Cox回归模型。该模型又英国统计学家D.R.Cox于1972年提出,主要用于肿瘤和其他慢性病的预后分析,也可用于队列研究的病因探索。Cox回归模型能处理多个因素对生存时间影响的问题。
这里用到的癌症是:宫颈鳞状细胞癌CESC(临床307个样本,基因表达有304个样本)
1.TCGA数据库下载宫颈鳞状细胞癌数据
首先需要合并差异基因得到的表达量和临床信息
这个步骤非常重要,也是让很多人感觉麻烦的地方,TCGA数据库样本量大,一个重要的癌症样本300-500个,临床信息又是独立存在,这里用到的是总生存时间和生存状态,得到一个行名是样本,列名包括总生存时间、生存状态、以及所有差异基因,对应的数据是差异基因的表达量,当然这个表达量是处理过的,不是TCGA下载下载下来的原始数据。
如果还没有得到生存时间、生存状态的文件,也没有得到差异基因的表达量,那就要先做差异分析,提取生存时间。简单回顾一下,提取生存时间会用到TCGA数据库下载的metadata.txt文件,这个文件大家很熟悉,可以直接在TCGA数据库下载的;差异分析涉及的内容就比较多,首先要从TCGA数据库下载基因表达数据,然后用perl脚本合并所有样本的表达矩阵,得到矩阵之后,要对ID进行转换,TCGA数据库用的是ensmbol ID,需要转换gene symobl,得到gene symobl的矩阵之后,就可以做差异分析,做了差异分析,就可以接着我们上面的合并工作了。
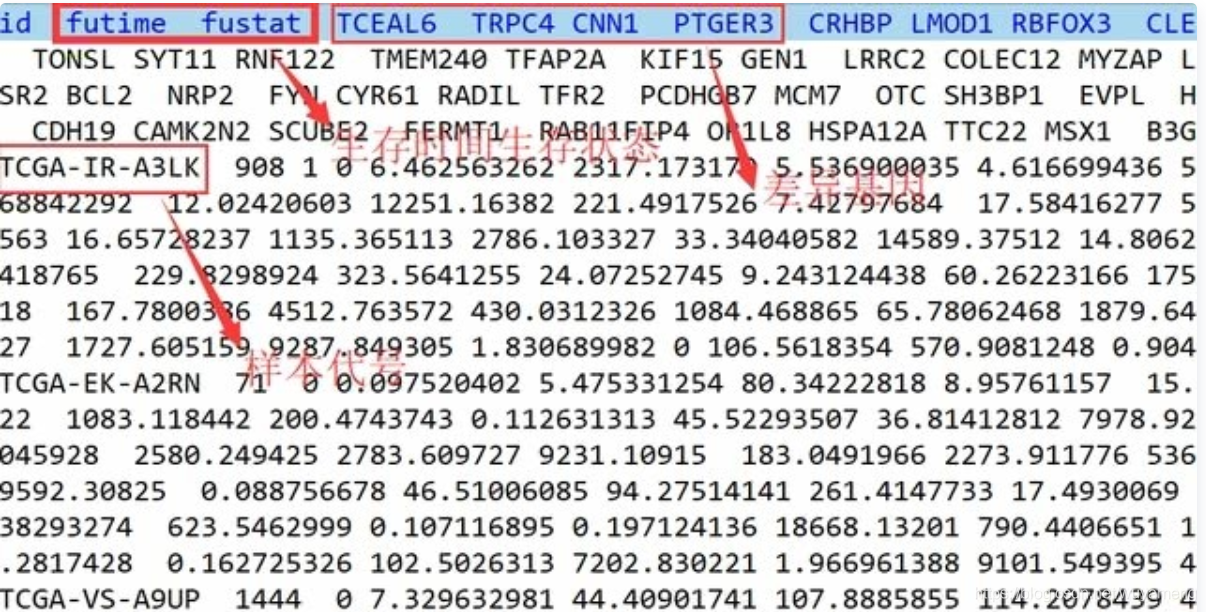
TCGA临床数据于表达数据合并
2.单因素Cox分析
有了生存时间和表达量合并的文件,就可以做单因素Cox分析,直接用我们的R做分析,得到这样一个表格文件。
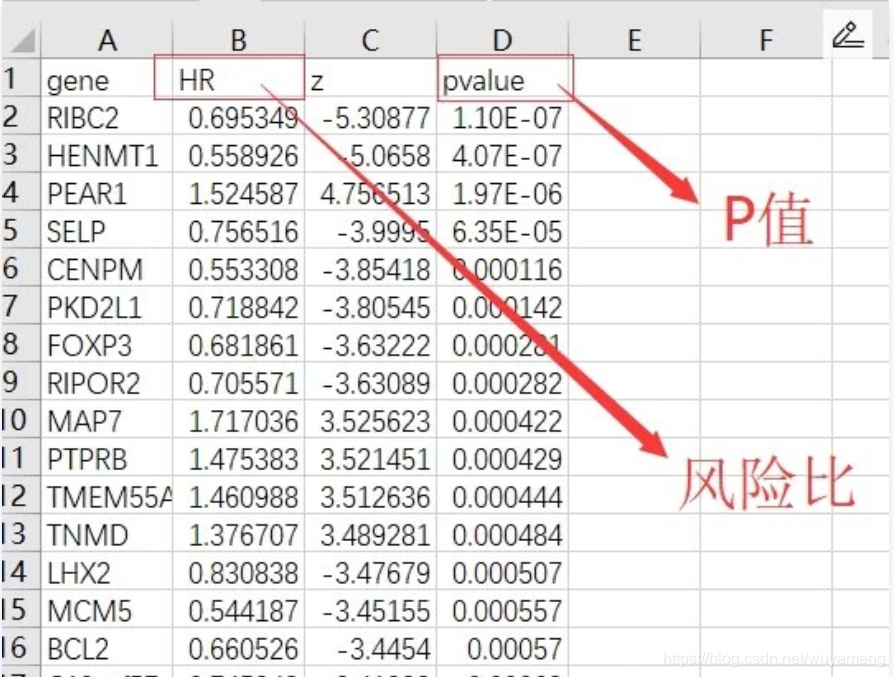
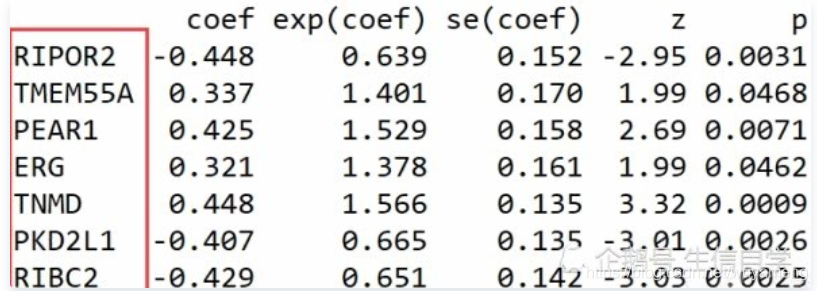
单因素cox分析
3.提取单因素P值
Cox单因素分析得到了单个基因的风险比和P值,可以筛选P值一个标准的基因,拿到这些基因,然后把这些基因的表达量筛选出来,还有样本的生存时间和生存状态,放在一个文件里面,用来做这些基因的多因素分析,当然了,筛选的基因不要多,控制在20个左右。简单点说,就是筛选这20个左右基因如同步骤一的文件。
4.多因素Cox分析
利用上面得到的关键基因的表达量做多因素分析,方法和单因素的差不多,只是这时用到了所有基因,而单因素是对每个基因做分析,多因素是用这些关键基因一起分析。可以得到风险值和高低风险分类。
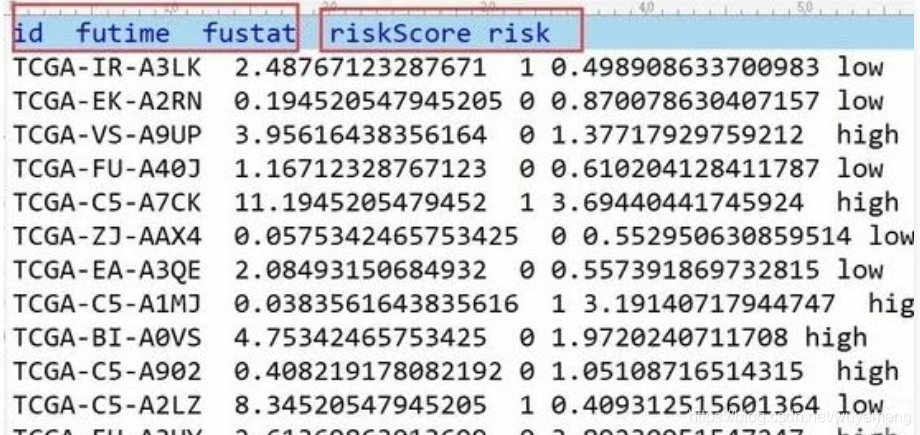
风险表格
5.绘制生存曲线、ROC曲线
用到的都是上面多因素分析得到的数据,用所有样本的风险比例,生存时间,就可以做生存曲线,ROC曲线。

风险生存曲线
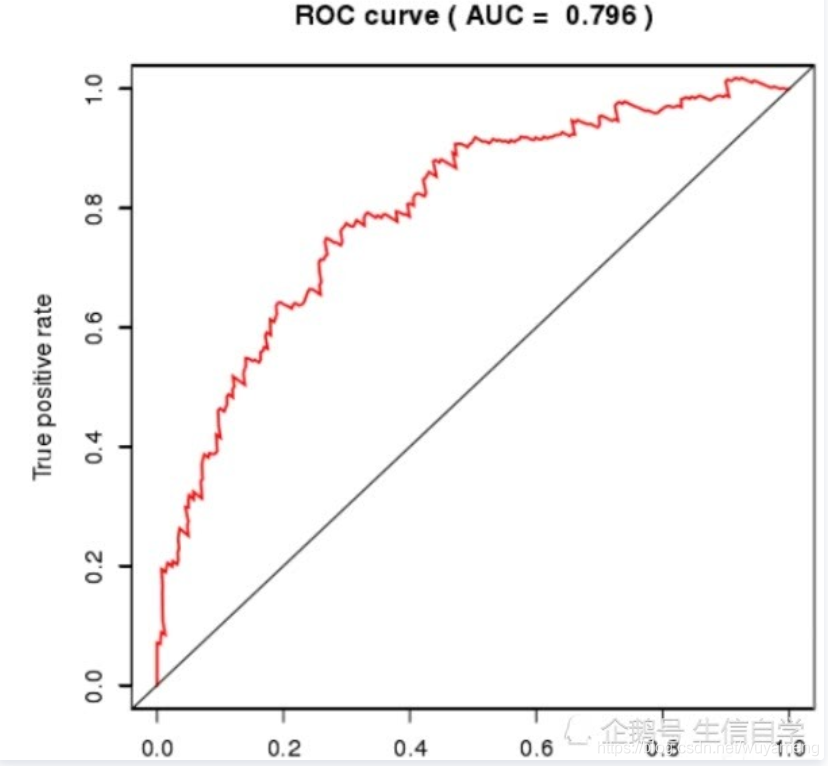
ROC曲线
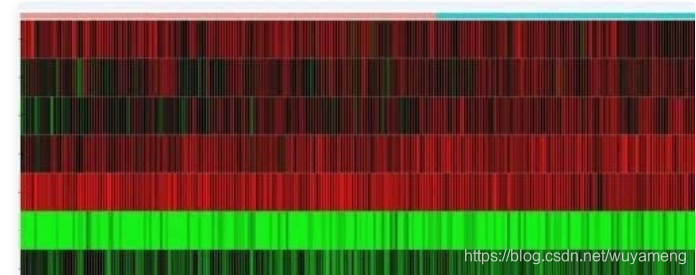
6.高低风险热图绘制
这里需要用到两个数据,一个是Cox多因素分析得到的基因,这个是根据Cox公式计算得到的,这里我们得到了7个,提取这7个基因的表达量,还有这7个基因在高低风险的分类,就可以绘制一张热图,热图从左到右的样本是风险分值以此从低到高的。
R语言实例练习:
单因素回归分析:
library("survival")
#install.packages('survminer')
library("survminer")
data("lung")
#Surv()函数创建生存数据对象(主要输入生存时间和状态逻辑值),再用survfit()函数对生存数据对象拟合生存函数
#Surv:用于创建生存数据对象
#survfit:创建KM生存曲线或是Cox调整生存曲线
fit <- survfit(Surv(time, status) ~ sex, data = lung)
#survdiff:用于不同组的统计检验
survdiff(Surv(time, status) ~ sex, data = lung)
#三种作图方法:
plot(fit)
ggsurvplot(fit,
pval = TRUE, conf.int = TRUE,
risk.table = TRUE, # Add risk table
risk.table.col = "strata", # Change risk table color by groups
linetype = "strata", # Change line type by groups
surv.median.line = "hv", # Specify median survival
ggtheme = theme_bw(), # Change ggplot2 theme
palette = c("#E7B800", "#2E9FDF")
)
plot(fit,xlab="Time(Days)",ylab="Survival",main="title",col=c("blue","red"),lty=2,lwd=2)
legend("topright",c("A","B"),col=c("blue","red"),lty=2,lwd=2,cex=0.7)
第一个作图语句输出:

Cox回归分析:
library("survival")
library("survminer")
#Cox模型主要用到的是coxph()函数,但需要先用Surv()函数产生一个生存对象;
#另外coxph()函数支持的方法有:exact,breslow以及exact,默认是exact
data("lung")
# res.cox <- coxph(Surv(time, status) ~ sex, data = lung)
# summary(res.cox)
#coef就是公式中的回归系数b(有时也叫做beta值)
#因此exp(coef)则是Cox模型中最主要的概念风险比(HR-hazard ratio)
# HR = 1: No effect
# HR < 1: Reduction in the hazard
# HR > 1: Increase in Hazard
# 在癌症研究中:
# hazard ratio > 1 is called bad prognostic factor
# hazard ratio < 1 is called good prognostic factor
res.cox <- coxph(Surv(time, status) ~ age + sex + ph.ecog, data = lung)
summary(res.cox)
# Create the new data
sex_df <- with(lung,
data.frame(sex = c(1, 2),
age = rep(mean(age, na.rm = TRUE), 2),
ph.ecog = c(1, 1)
)
)
fit <- survfit(res.cox, newdata = sex_df)
ggsurvplot(fit, data = sex_df, conf.int = TRUE,
legend.labs=c("Sex=1", "Sex=2"),
ggtheme = theme_minimal())
