基因表达
什么是基因表达,如下是来自于维基百科的解释:
Gene expression
is the process by which information from a
gene
is used in the synthesis of a functional
gene product
. These products are often
proteins
, but in non-protein coding genes such as
transfer RNA (tRNA)
or
small nuclear RNA (snRNA)
genes, the product is a functional
RNA
. The process of gene expression is used by all known life—
eukaryotes
(including
multicellular organisms
),
prokaryotes
(
bacteria
and
archaea
), and utilized by
viruses
—to generate the
macromolecular
machinery for life.

研究方法
定量PCR
这部分我不太懂,所以就放几段百度百科和维基百科的定义。
- 百度百科
定量PCR(即时聚合酶链锁反应,Real-time Polymerase Chain Reaction,简称 Real-time PCR、即时PCR),又称定量即时聚合酶链锁反应(Quantitative real time polymerase chain reaction,简称 Q-PCR/qPCR/rt-qPCR、定量即时PCR、即时定量PCR),是一种在DNA扩增反应中,以萤光染剂侦测每次聚合酶链锁反应(PCR)循环后产物总量的方法技术,有广义概念和狭义概念。广义概念的定量PCR技术是指以外参或内参为标准,通过对PCR终产物的分析或PCR过程的监测,进行PCR起始模板量的定量。狭义概念的定量PCR技术(严格意义的定量PCR技术)是指用外标法(荧光杂交探针保证特异性)通过监测PCR过程(监测扩增效率)达到精确定量起始模板数的目的,同时以内对照有效排除假阴性结果(扩增效率为零)。
- 维基百科
A real-time polymerase chain reaction (Real-Time PCR), also known as quantitative polymerase chain reaction (qPCR), is a laboratory technique of molecular biology based on the polymerase chain reaction (PCR). It monitors the amplification of a targeted DNA molecule during the PCR, i.e. in real-time, and not at its end, as in conventional PCR. Real-time PCR can be used quantitatively (quantitative real-time PCR), and semi-quantitatively, i.e. above/below a certain amount of DNA molecules (semi quantitative real-time PCR).
优点:灵敏性高,准确性高,通量也还行。一般而言,RNA-Seq和microassay分析得到的差异表达基因最终也需要通过这种实验方法进行验证。
但是一般适用于验证实验,而不是用于探索性实验。
microarray<small>基因矩阵</small>
基因芯片的概念在上个世纪80年代就已经提出来了, 被评为1998年度自然科学领域十大进展之一。他的基本原理通过设计专门的短核苷酸作为探针,把这些探针固定在专门的基片表面,然后用样本的cDNA进行杂交,根据杂交信号的强弱来判断基因表达的程序。
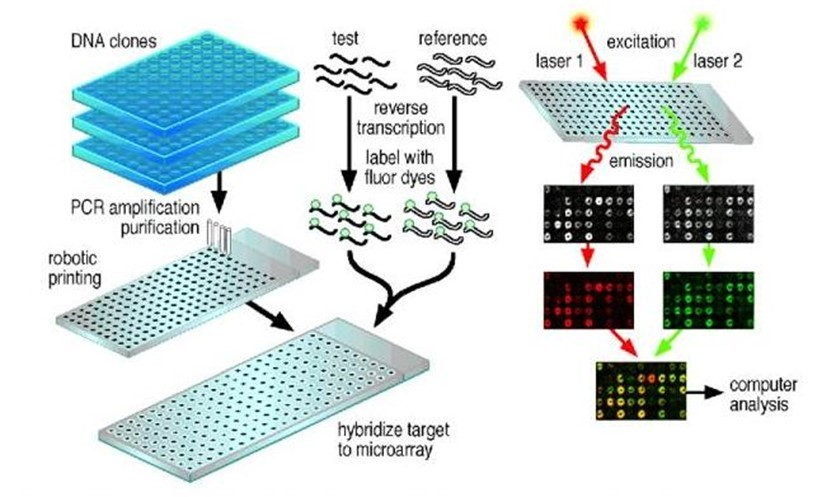
但是microarray检测的基因数量完全取决于你的探针设计的数量,而且难以研究mRNA的可变剪切。
RNA-Seq
RNA-Seq是目前基因表达分析最常用的技术。分为以下几步
- 分离所有mRNA
- 逆转录mRNA成cDNA
- 对cDNA测序
- 比对参考基因组
RNA-Seq实验设计中的“重复”包括:技术重复和生物学重复
重复是为了检测组间和组内的变异,对于假设检验至关重要。
- 技术重复为了估计测量技术(RNA-Seq)的变异。
-
生物学重复是为了发现生物组内的变异。
简单的说,两组的基因表达的变化只有比组内变异还大时才能认为时显著的。
RNA-Seq的概率分布
相同基因在不同细胞的表达水平服从log-normal(对数正态)分布,由定量PCR验证。(
注
:这与相同细胞不同基因表达的分布不同)但是大多数基因表达实验都是用一群细胞,几乎没有相应分布提出。
RNA-Seq试验中,抽样得到的raw read counts服从泊松分布。并且同一样本在两次试验中的结果不同,这称为shot noise。这种变异在RNA-Seq
技术重复
间成为Possion noise。
生物学上不同的样本间的差异服从负二项(negative binomial)分布,有时称gamma-Poisson分布。
由于RNA-Seq count数据也表现出zero inflation(大量值为0)的特征,所以很难拟合到负二项分布,所以有文章认为要用Poisson-Tweedie family建模。
RNA-seq数据和microassay在差异表达分析上的区别:
-
RNA-Seq观察到的数据是抽样过程中产生的离散(discrete)count形式。也就是说总体是恒定的,表达量越高的基因在抽样结果中所占的比例越大。表达量低的基因可能即便有也无法被检测出来。当然,重新对相同文库进行测序,还是有可能找到更多表达的转录本
-
microassay检测的是荧光信号的连续度量。由于使用固定的核酸序列去杂交,所以不是一种“零和游戏”,只要能杂交,就能被检测。(但如果没有设计相应的引物,就不能检测到可能的基因)
研究意义
1.在不同背景下比较mRNA水平
- 同一物种,不同组织:研究基因在不同部分的表达情况
- 同一物种,同一组织:研究基因在不同处理下,不同条件下的表达变化
- 同一组织,不同物种:研究基因的进化关系
- 时间序列实验: 基因在不同时期的表达情况与发育的关系
2.基因